優點:
計算速度快
模型運算方式較容易理解
缺點:
使用平均值的算法容易受到離群值的影響
對於類別性變數並不適用平均數,因此僅適用於數值型變數
各類樣本數差異較大時,容易產生錯誤分類
K-Medoids和K-Means的方式類似,步驟如下:
K-Medoids和K-Means的差異僅有兩處,分別為中心點的選擇(K-means:虛擬點v.s K-medoids實際樣本點)和中心點決定的方式(K-Means:群內樣本的座標平均值v.s K-medoids: 群內距離和最小的樣本點)。由於決定中心點的方式不同,因此K-Medoids較不容易受到離群值的影響,且可以處理類別型資料,但運算量相對而言較大。
stats套件中的kmeansset.seed(123)
# 建立模型,一開始假設nstart群
kmeans_model <- kmeans(testing[,1:12], centers = 13, nstart = 25,iter.max = 300)
# 找出分群結果
pred_kmeans <- as.factor(kmeans_model$cluster-1) ## activity 起始值為0
# 衡量
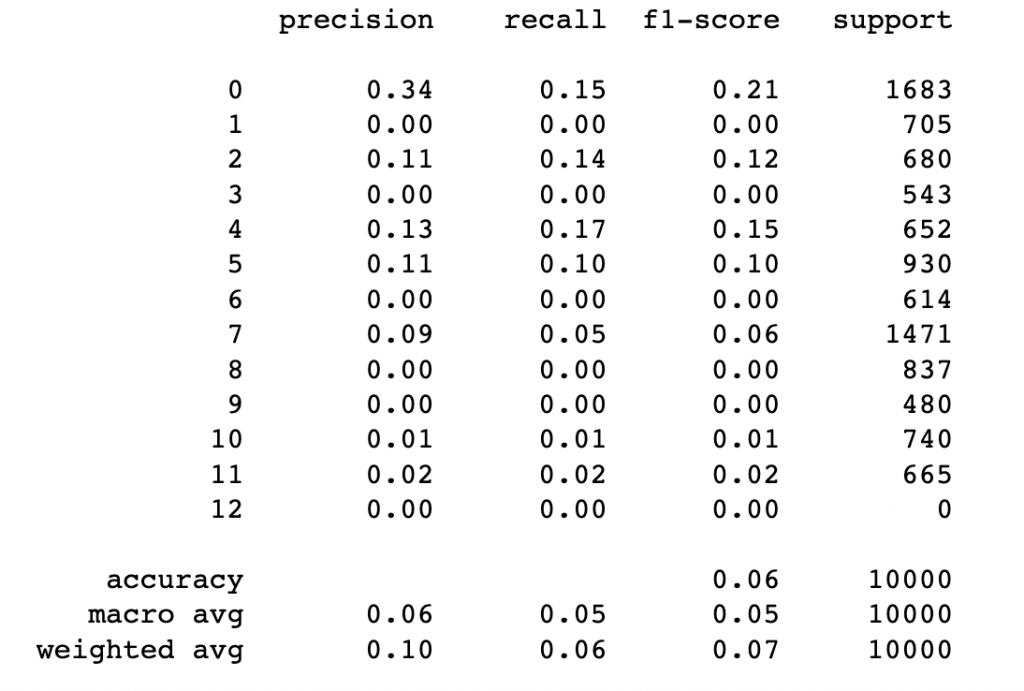
confusionMatrix(pred_kmeans,reference = as.factor(testing$Activity))
根據混淆矩陣可知,kmeans的結果並不好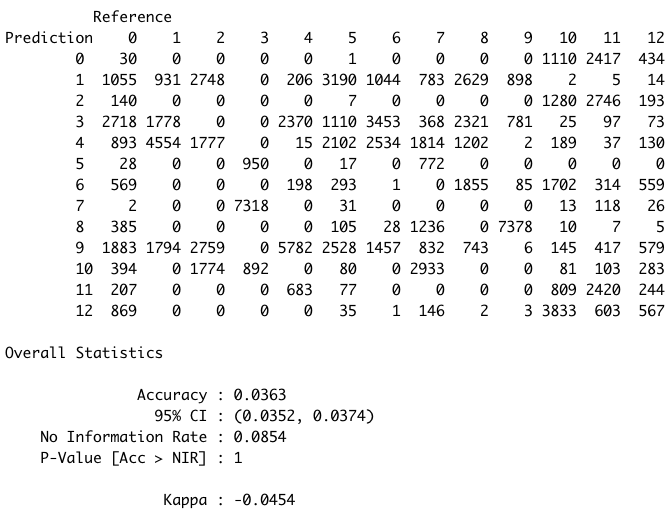
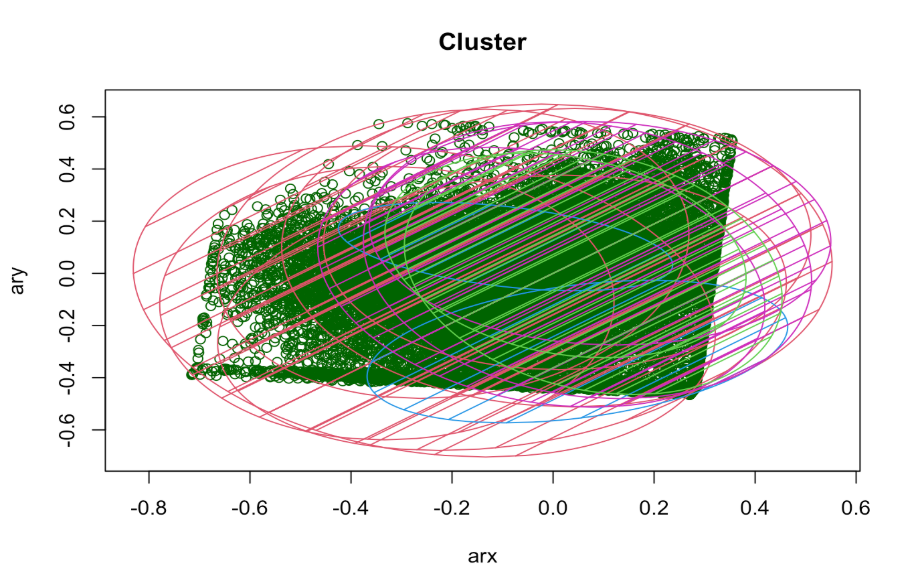
## 視覺化結果,使用其中一個加速度計的xy軸數值,作為視覺化呈現
library(cluster)
y_kmeans <- kmeans_model$cluster-1
clusplot(testing[, c("arx", "ary")],y_kmeans,lines = 0,
shade = TRUE,color = TRUE,
labels = 2,plotchar = FALSE,
span = TRUE,
main = paste("Cluster"),
xlab = 'arx',
ylab = 'ary')
sklearn.cluster套件中的KMeansfrom sklearn import cluster
# 建立模型
pred_kmean = cluster.KMeans(n_clusters = 13,algorithm="elkan")
# 預測分群結果
pred_kmean.fit_predict(X_test)
pred_labels = pred_kmean.labels_
# 衡量結果
from sklearn import metrics
print(metrics.classification_report(pred_labels, Y_test))
根據混淆矩陣可知,kmeans的分群結果並不好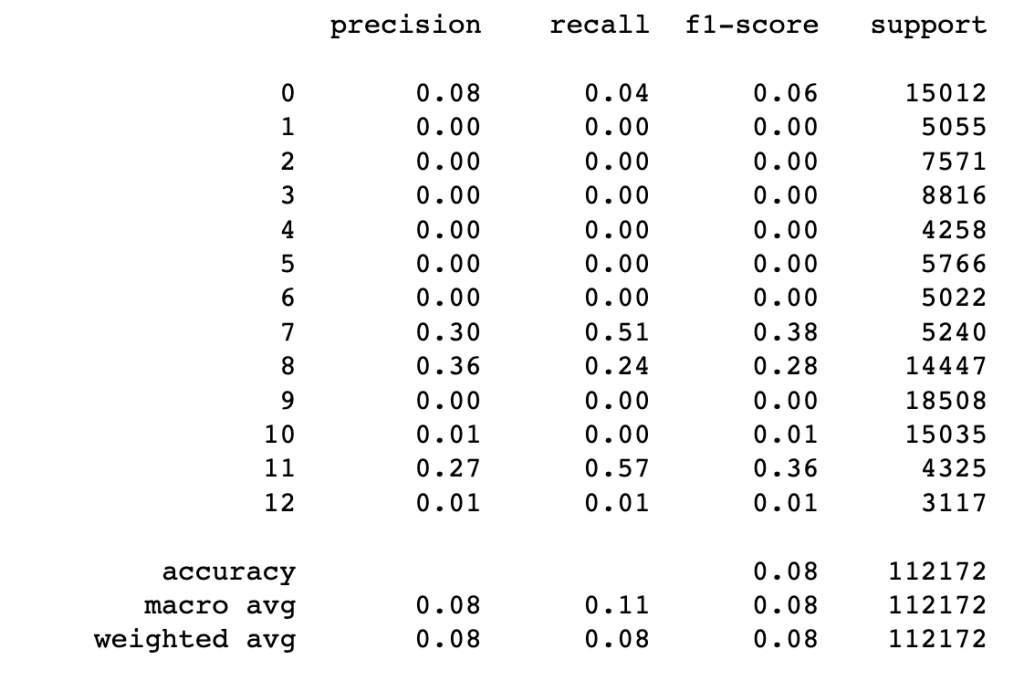
cluster套件中的pamlibrary(cluster)
# 為了減小計算量,因此隨機在testing set中抽取了10000筆資料來做示範
subset_testing <- testing[sample(1:length(testing$Activity),10000),]
# 模型
model_kmedoids <- pam(subset_testing[,1:12], 13)
# 預測分群結果
pred_kmedoids <- as.factor(model_kmedoids$clustering-1)
# 衡量結果
confusionMatrix(pred_kmedoids,reference = as.factor(subset_testing$Activity))

sklearn_extra.cluster套件中的KMedoidsfrom sklearn_extra.cluster import KMedoids
# 為了減小計算量,因此隨機在testing set中抽取了10000筆資料來做示範
index = list(range(0,(X_test.shape[0])-1))
sample_index = random.sample(index, 10000)
# 模型
pred_KMedoids = KMedoids(n_clusters=12, random_state=0).fit(X_test[sample_index,:])
# 印出分群結果
pred_labels = pred_KMedoids.labels_
# 衡量結果
print(metrics.classification_report(pred_labels, Y_test.iloc[sample_index]))